Laboratoire
Test qPCR avec transcriptase inverse : SARS-CoV-2 dans les échantillons d’eaux usées
avr. 1 2021
Bureau Veritas offre un test permettant de quantifier le virus SARS-CoV-2 dans les échantillons d’eaux usées. Ce test utilise la méthode de réaction en chaîne par polymérase avec transcriptase inverse (RT-qPCR) en une étape.
Le virus du SARS-CoV-2 se déplace principalement vers les solides, plutôt que vers la phase aqueuse. En ciblant les solides dans un échantillon d’eau usée, le test fournit un résultat plus sensible et plus représentatif.
Prélèvement et conservation des échantillons
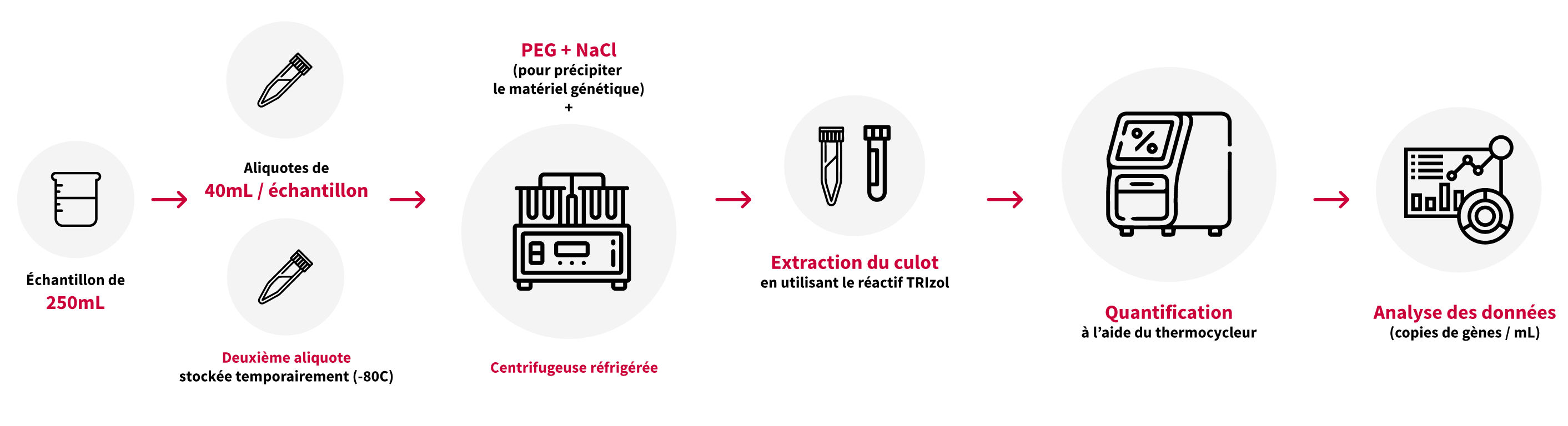
Les échantillons sont prélevés dans des contenants de 250 mL sans agent de conservation. Les échantillons doivent être immédiatement réfrigérés et le traitement en laboratoire doit commencer dans les 48 heures suivant le prélèvement.
Le matériel génétique du virus SARS-CoV-2 a tendance à se dégrader dans les eaux usées au cours des 48 heures suivant le prélèvement, s’il est conservé à température ambiante. La réfrigération des échantillons d’eaux usées peut minimiser la perte de matériel génétique pendant 4 à 5 jours.
Préparation et extraction des échantillons
Le contenu de chaque contenant de 250 mL est homogénéisé. Deux aliquotes de 40 mL sont versées dans des tubes séparés. Une aliquote est traitée pour l’analyse, tandis que la seconde est conservée dans un congélateur, au cas où l’échantillon nécessiterait une nouvelle analyse.
L’aliquote de 40 mL soumise au laboratoire est traitée à l’aide de polyéthylène glycol (PEG) et de chlorure de sodium (NaCl) pour précipiter le matériel génétique en suspension. Les tubes sont centrifugés à 4 °C en deux étapes successives, en éliminant autant de surnageant que possible et en soumettant les solides résultants (culot) à un traitement ultérieur.
Bureau Veritas utilise la méthode d’extraction TRIzol™ pour isoler le matériel génétique du culot. Une fois extrait, le matériel génétique est stable pendant de longues périodes s’il est congelé.
La figure 1, ci-dessous, présente un résumé de la méthode de Bureau Veritas pour quantifier le matériel génétique du SARS-CoV-2 dans les échantillons d’eaux usées.
Figure 1. Résumé de la méthode Bureau Veritas pour le dépistage du SARS-CoV-2 dans les échantillons d’eaux usées : préparation de l’échantillon, extraction et quantification.
Détection et quantification par PCR
La RT-qPCR est une méthode bien établie pour identifier et quantifier l’acide désoxyribonucléique (ADN) double brin. Le virus SARS-CoV-2 contient un acide ribonucléique (ARN) à brin unique, qui nécessite une transcription inverse en ADN complémentaire (ADNc). La méthode de Bureau Veritas combine la transcription inverse, l’amplification par PCR de l’ADNc obtenu et la quantification en une seule étape. Elle est donc appelée méthode qPCR en 1 étape. Cette approche offre une meilleure sensibilité et des résultats plus représentatifs.
L’identification et la quantification du matériel génétique par PCR ciblent les gènes qui sont spécifiques à un pathogène particulier. Dans ce cas, la méthode de Bureau Veritas identifie et quantifie les fragments N1 et N2 du gène N du SARS-CoV-2, qui code pour la protéine de la nucléocapside du virus (domaines N1 et N2).
Outre les fragments N1 et N2 du SARS-CoV-2, chaque échantillon est également analysé pour le virus de la marbrure légère du piment (PMMoV), en tant que biomarqueur du contenu fécal. Plus important encore, afin de minimiser la variabilité des données inhérente aux échantillons d’eaux usées, la moyenne des copies de gènes par mL pour le SARS-CoV-2 (N1 et N2 combinés) est normalisée par rapport aux copies de gènes par mL du PMMoV.
Rapport des données
Le certificat d’analyse rapporte la présence ou l’absence du virus SARS-CoV-2 sur la base de l’amplification des gènes cibles (N1 et N2) en 40 cycles PCR. Les données quantitatives sont rapportées en tant que copies de gènes par mL de chacun des gènes cibles ainsi qu’en tant que moyenne des copies de gènes par mL des N1 et de N2. La limite de détection de la méthode est de 2 copies de gènes/mL. L’amplification du matériel génétique du virus au-delà de 40 cycles PCR est considérée comme « présente mais non quantifiable ».
Utilité des données
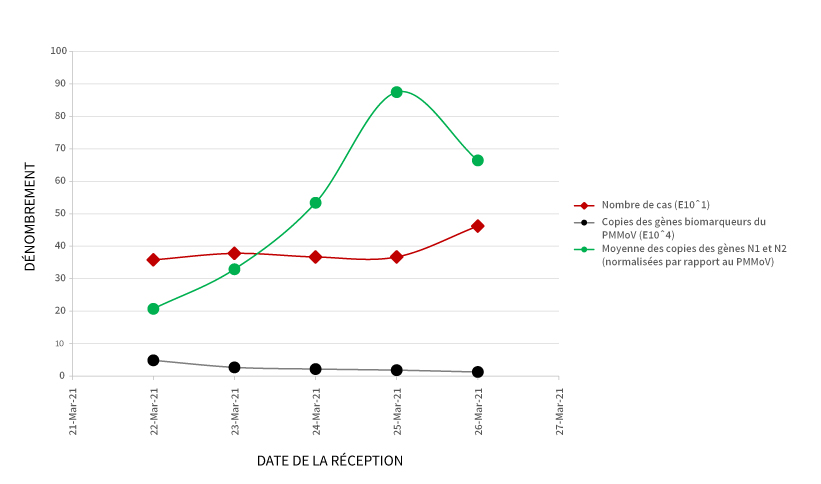
À titre d’exemple de l’utilisation de ces résultats, la figure 2 (ci-dessous) présente les tendances des données provenant d’une usine de traitement du sud de l’Ontario. En plus des copies de gènes normalisées par mL, le graphique comprend le nombre de cas associés signalés par l’autorité locale de santé publique et les copies de gènes par mL du biomarqueur PMMoV.
Figure 2. Évolution du matériel génétique du SARS-CoV-2 (données normalisées) dans les échantillons d’eaux usées, par rapport au nombre de cas et au biomarqueur PMMoV.